Trichoderma, loài nấm tơ trong đất, có khả năng ký sinh trên nhiều loài nấm gây bệnh trên cây. Mười hai dòng phân lập Trichoderma spp thu từ các địa phương khác nhau ở Nam Andaman được mô tả quy trình nuôi cấy, hình thái học và hoạt tính kháng lại các bệnh ở phần trong đất và lá. Các trình tự dòng phân lập cho thấy 7 loài khác nhau. Các dòng phân lập cho thấy các mẫu phản ứng khác nhau với các kiểm tra bệnh như Sclerotium rolfsii, Colletotrichum gloeosporioides và C. capsici.
Tóm tắt:
Trichoderma, loài nấm tơ trong đất, có khả năng ký sinh trên nhiều loài nấm gây bệnh trên cây. Mười hai dòng phân lập Trichoderma spp thu từ các địa phương khác nhau ở Nam Andaman được mô tả quy trình nuôi cấy, hình thái học và hoạt tính kháng lại các bệnh ở phần trong đất và lá. Các trình tự dòng phân lập cho thấy 7 loài khác nhau. Các dòng phân lập cho thấy các mẫu phản ứng khác nhau với các kiểm tra bệnh như Sclerotium rolfsii, Colletotrichum gloeosporioides và C. capsici. Tuy nhiên, các dòng phân lập, TND1, TWN1, TWC1, TGD1 và TSD1 ức chế sự tăng trưởng sợi nấm hiệu quả nhất của các bệnh được kiểm tra. Hoạt tính chitinase và β-1,3-glucanase đáng kể của tất cả các Tricoderma phân lập được ghi nhận trong môi trường tăng trưởng. T. viride được phát hiện với hoạt tính chitinase cao nhất trong khi T. harzianum có ghi nhận với hoạt tính β-1,3-glucanase cao nhất..
Từ khóa: Trichoderma, Spice, Biocontrol, Andaman and Nicobar Islands
Giới thiệu
Chi nấm sợi Tricoderma (Hypocreales, Ascomycota) là loài nấm trong đất có ở khắp nơi, thường được tìm thấy trong gỗ mục [1,2] và một số loài cho sản phẩm có giá trị kinh tế như các enzyme công nghiệp (Trichoderma ressei) [3], kháng sinh [4] và được dùng như các tác nhân kiểm soát sinh học (ví dụ T. harzianum, T. atroviride and T. asperellum) chống lại các bệnh trên cây trồng [5]. Tricoderma spp thường được phân lập trong đất và trong hệ thống rễ [6]. Tuy nhiên, nhiều cân nhắc trong tìm ra nhiều nấm mycoparasitic đặc biệt hơn trong Trichoderma spp., vốn khác biệt khi liên hệ với hoạt tính kiểm soát sinh học. Điều này quan trọng để phân lập Trichoderma spp có tiềm năng hiệu quả kháng từ chọn lọc các dòng phân lập có tiềm năng cao thêm vào các enzyme lytic tế bào chitinase và β-1,3-glucanase. Các enzyme lytic phá vỡ thành tế bào polysaccharides thành các oligomers ngắn tạo điều kiện hyperparasite xâm nhập vào tế bào chất của nấm mục tiêu [7]. Mục đích của nghiên cứu này là kiểm tra Trichoderma spp cho khả năng kháng bằng nuôi cấy kép cũng như khả năng sản xuất enzyme lytic đối kháng các bệnh.
Vật liệu và phương pháp nghiên cứu
Mẫu đất và phân lập
Mẫu đất được thu thập từ nhiều môi trường sinh thái của cây ớt của quận South Andaman, Andaman và Nicobar Islands (India) để phân lập Tricoderma spp. (Bảng 1). Mẫu được đem về phòng thí nghiệm và ủ ở 4°C đến khi dùng. Mỗi mẫu được pha loãng 5 lần liên tiếp được chuẩn bị với nước cất vô trùng và 0.5mL mẫu pha loãng được đổ lên bề mặt của môi trường Trichoderma Specific Medium (TSM) [8]. Các đĩa được ủ ở 28 ± 2°C trong 96h. Hình thái học khác nhau của khuẩn lạc xuất hiện trên thạch được thuần chủng hóa trong Potato Dextrose Agar (PDA) (HiMedia, India). Các dòng thuần được bảo quản ở 4°C và dùng trong suốt quá trình nghiên cứu.
Đặc điểm hình thái của các chủng Trichoderma
Hình thái học và đặc điểm nuôi cấy của 12 chủng Tricoderma được nghiên cứu trên 4 môi trường khác nhau bao gồm OMA, CMD, PDA và TSM theo quy trình của Samules và cs [9]. Đĩa nấm sợi (6mm) của tiền nuôi cấy của các dòng Tricoderma tương ứng được nuôi cấy ngoại vi trên đĩa petri chứa các môi trường nói trên và ủ ở 28 ± 2°C trong 1 tuần. Bán kính khuẩn lạc được đo sau 24, 48 và 72 giờ. Mỗi thí nghiệm mức tăng trưởng được lặp lại 3 lần và kết quả được tính trung bình cho mỗi dòng. Các đặc tính bổ sung bao gồm sự hiện diện của sắc tố, bào tử nguyên phân xanh, mùi hôi và xuất hiện khuẩn lạc được ghi nhận.
Các quan sát hình thái học được ghi nhận từ các nuôi cấy tăng trưởng trên các đĩa PDA. Các đặc tính sau được tính toán; chiều rộng thể bình ở pint rộng nhất, chiều dài thể bình, chiều dài và chiều rộng bào tử nguyên phân và sự xuất hiện của bào tử áo. Mỗi đặc điểm được đo từ trong nước sau khi làm ướt với KOH 3% cho mỗi chủng phân lập.
Điều kiện tăng trưởng nấm và ly trích DNA
Nuôi cấy được duy trì trên PDA ở 25°C được tăng trưởng trên potato dextrose broth trong 3 ngày. Lớp tơ nấm được thu nhận trên giấy lọc, rửa vưới nước cất 2-3 lần, đông khô và được dùng để ly trích DNA. Bộ gen DNA được ly trích bằng phương pháp của Raeder và Broda [10]. DNA huyền phù trong 50µl đệm TE và định lượng bằng ethidium bromide fluorescence.
Khuếch đại PCR và giải trình tự sản phẩm khuếch đại
Mồi ITS1 (5’-TCC GTA GGT GAA CCT GCG G-3’) và ITS4 (5’-TCC TCC GCT TAT TGA TAT GC-3’) được mô tả bởi White và cs [11] được dùng để khuếch đại đoạn rDNA bao gồm ITS và ITS và gen 5.8S rDNA. Khuếch đại PCR được thực hiện trong tổng thể tích 50 µl bằng cách mix 20ng của DNA mẫu với nồng độ 2.5 mM của mỗi deoxynucleotide triphosphate, nồng độ 1 µl mỗi mồi và 3 U của Taq DNA polymerase trong 109 Taq buffer A (GeNeI). Các phản ứng này được biến tính trong 1 phút ở 95°C, sau đó thực hiện 35 chu kỳ 1 phút ở 95°C, 30s ở 55°C và 1.5 phút ở 72°C, giai đoạn cuối ở 72°C trong 10 phút sử dụng GeneAmp PCR system 9700 (Applied Biosystems). Các sản phẩm PCR được điện di bằng gel agarose 1%. Trình tự của sản phẩm PCR tinh khiết được thực hiện bằng thiết bị SAP, GeneI, Banglore sử dụng ITS1 (forward primer).
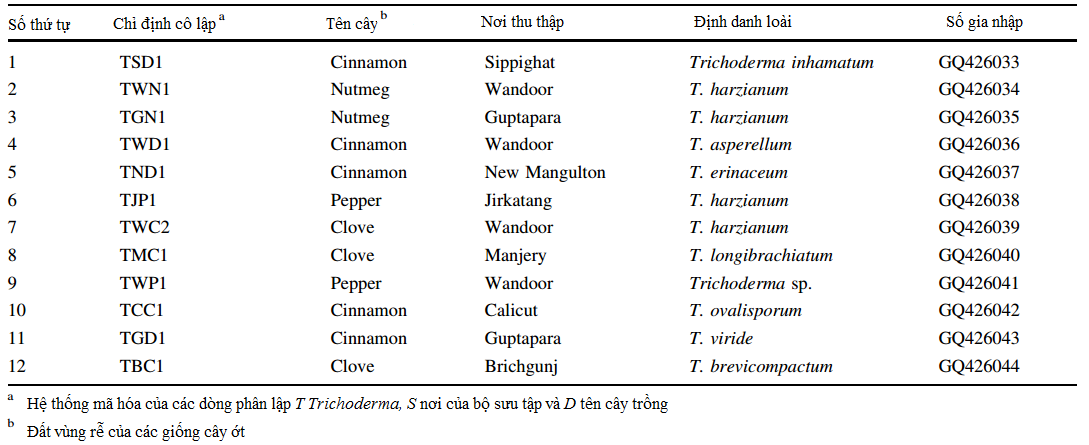
Bảng 1
Phân tích phát sinh loài
Các trình tự gen ITS của các dòng được so sánh với các trình tự có sẵn bằng BLAST tìm kiếm trên NCBI, dữ liệu GenBank (http://www.ncbi.nlm.nih.gov). Sự sắp xếp nhiều trình tự được thể hiện bằng Clustal X (1.1). Phương pháp của Jukes và Cantor [12] được dùng để tính khoảng cách tiến hóa. Phát sinh loài dendogram được xây dựng bằng phương pháp cây phát sinh loài và cây liên kết được đánh giá bằng phân tích thực hiện bootstrap của 100 set dữ liệu sử dụng MEGA 3.1 (Molecular Evolutionary Genetic Analysis). Các trình tự được gửi đến GenBank dưới số gia nhập sau GQ426033–GQ426044 (12 trình tự).
Hoạt tính đối kháng của các chủng Tricoderma
Kỹ thuật nuôi cấy kép mô tả bởi Morton và Stroube [13] được dùng trong kiểm tra tính đối kháng của Tricoderma spp. Chống lại tác nhân gây bệnh trong đất S. rolfsii và các tác nhân gây bệnh từ lá như C. capsici và C. gloeosporioides. Các tác nhân gây bệnh và Tricoderma được nuôi cấy trên PDA trong 1 tuần ở nhiệt độ phòng (28± 2oC). Các khối nhỏ của nấm mục tiêu (C. capsici và C. gloeosporioides) cắt từ vùng ngoại biên được chuyển sang đĩa petri trước khi rót PDA. Sau hai ngày Colletotrichum spp. tăng trưởng, Tricoderma spp. được chuyển trong điều kiện vô trùng sang đĩa tương ứng và được ủ ở nhiệt độ phòng với chu kỳ sáng tối trong 7 ngày và được quan sát định kỳ. Cả bệnh S. rolfsii và Tricoderma được ủ trong cùng thời gian. Thí nghiệm được lặp lại ba lần và phần trăm ức chế tăng trưởng được đo bằng công thức I = (C-T)/C 9 100, với C là tăng trưởng tơ ở đĩa đối chứng, T là sự tăng trưởng tơ trong đĩa nuôi kiểm tra vi sinh vật và I là sự ức chế sự tăng trưởng của tơ nấm.
Thử nghiệm hoạt tính enzyme của dịch chiết tế bào
Mười hai dòng phân lập của Trichoderma được tiêm riêng vào 100 ml môi trường nuôi cấy với bốn sự kết hợp, gồm Czapek Dox Broth riêng lẻ, CDB + bột tơ nấm S.rolfsii (10 g), CDB + bột tơ nấm của C.gloeosporioides (10g) và CDB + bột tơ nấm C.capsici (10g) trong bình Erlenmeyer và ủ ở 28 ± 1°C trong 7 ngày trong điều kiện liên tục ở tốc độ 125 vòng/phút hai lần một ngày. Dịch nuôi cấy được lọc ở mỗi dòng cô lập được thu, lọc qua giấy lọc giấy lọc Whatman 42, ly tâm và khảo sát các hoạt động của chitinase và β -1,3-glucanase ngay lập tức.
Hoạt tính β-1,3-glucanase
Phân tích enzyme β-1,3-glucanase, 0.5 ml laminarin, 1.0 ml của 0.05 M đệm citrate (pH 4.8) và 0.5 ml dịch lọc được mix và nuôi ở 40°C trong 60 phút. Thuốc thử dinitrosalicyclic acid được thêm vào hỗn hợp phản ứng với thể tích bằng nhau và làm ấm trong nước đun sôi trong 15 phút. Sự hấp thụ của hỗn hợp phản ứng được đo ở 575 nm trong máy đo quang phổ và so sánh với đồ thị đường chuẩn có quy trình tương tự nhưng sử dụng các nồng độ glucose khác nhau thay vì dịch lọc. Định lượng lượng đường sụt giảm được tính từ đường glucose chuẩn dùng trong phân tích hoạt tính β-1,3-glucanase có đơn vị là nkat/ml. Một nkatal tương ứng với sự giải phóng 1nmol glucose trong 1 giây.
Hoạt tính chitinase
Hỗn hợp 0.5ml dịch lọc, 0.5 ml huyền phù keo chitin và 1.0 mL đệm Mcllvaines (pH 4.0) được mix và ủ ở 37°C trong 2 giờ trong bể nước lắc liên tục. Phản ứng dừng khi đun 3 phút trong bể nước gia nhiệt. 3ml thuốc thử kali ferricyanide được thêm và làm ấm trong nước sôi trong 15 phút. Lượng N-acetyl glucosamine (NAG) giải phóng được ước tính dựa theo phương pháp của Reissing và cộng sự [14]. Sự hấp thu của dịch phản ứng được đo bằng máy quang phổ ở bước sóng 420 nm. Phần lượng đường sụt giảm được tính từ đường cong chuẩn cho hoạt tính enzyme NAG và chitinase có đơn vị pkat (pmol/s) trên millitre.
Phân tích thống kê
Phân tích thống kê được thực hiện với Agres và Agdata sử dụng phân tích sai biệt hoàn toàn ngẫu nhiên (ANOVA) được dùng để so sánh hiệu quả kiểm soát sinh học của các chủng Trichoderma và giá trị có nghĩa được phân biệt bởi kiểm tra sự khác biệt có nghĩa ít nhất (LSD) của Fisher. Ảnh hưởng của các đặc tính tăng trưởng ở Trichoderma được đánh giá bằng độ lớn của giá trị F (P=0.05).
Kết quả và thảo luận
Đặc điểm hình thái học
Dựa vào quan sát của bào tử nguyên phân, bào tử dính, cấu trúc khuẩn lạc, bào tử vách dày, hình thái bào tử nguyên phân các chủng được phân nhóm thành Pachybasium B (nhánh Lixii/catoptron), Trichoderma (Rufa), Trichoderma (Pachybasium A), Longibrachiatum and Pachybasium B (Lutea). Phần Pachybasium bao gồm các phân lập TSD1, TWN1, TGN1, TJP1 and TWC1 nơi mà bào tử được phân nhánh nhiều, nảy sinh trong vòng khác biệt và duy trì như các khu vực. Các nhánh chính hầu hết trong nhóm 2-3 và nảy ra vuông góc và chiều dài của chúng tăng theo khoảng cách từ đỉnh của nhánh chính có sự xuất hiện hình nón hay kim tự tháp . Trong phần Tricoderma (Rufa) (TWD1, TGD1 and TCC1) các sợi nấm, phân nhánh, mịn và không màu. Các bào tử nguyên phân được quan sát cho thấy ít phức tạp và hình thành hyphae trên không khí. Chúng tạo ra các nhánh nhỏ hơn và cuối cùng một hệ thống phân nhánh hình cây tùng được hình thành. Trong Pachybasium B (Lutea) (TBC1) các bào tử nguyên phâns tạo vách mịn, nhánh thẳng theo kiểu Pachybasium. Trichoderma (phần của Pachybasium A) bao gồm T. asperellum quan sát thấy các bào tử nguyên phâns đã chấm dứt 2 hoặc nhiều philides và sự phân nhánh ban đầu phát sinh gần 90 độ so với trục chính. Trong phần Longibrachiatum (H. orientalis), các bào tử nguyên phân phức tạp và dài ra từ từ, thường kết hợp, nhiều nhánh thứ hai. Các thể bình phát sinh trực tiếp từ các nhánh thứ phát, thường không có dạng xoắn. Sử dụng ghi chú của Rifai [15] trong hầu hết các chủng được phân lập ở những vùng này được xác định là T. harzianum. T. hamatum và T. viride [16]. Trên thực tế, ngoài T. harzianum thường chiếm đa số các phân lập, một phát hiện thú vị của nghiên cứu này là sự tồn tại của một số loài Trichoderma (như T. harzianum, T. erinaceum, T. Asperellum, T. ovalisporum, T. viride và T. brevicompactum) trong hệ sinh thái đảo Bay.
Định danh phân tử các loài Tricoderma
Các phân tích trình tự của 12 dòng phân lập được thực hiện để xác nhận định danh loài được thực hiện trước đó chỉ dựa vào các thông số hình thái học. So sánh các đoạn mã hóa oligonucleotide của các chuỗi rDNA, bao gồm gen 5.8S và các vùng ITS1 và ITS2 bên cạnh, với các chuỗi tham khảo từ các cơ sở dữ liệu công bố, cho thấy rằng chúng tương tự nhau.
Phân tích phát sinh loài
Cây phát sinh loài thu được bằng cách phân tích trình tự của ITS1 và ITS4 trong số 12 trình tự các dòng phân lập và trình tự của 22 Trichoderma spp. thu được từ NCBI, GenBank được thể hiện trong Hình 1. Trình tự ITS đã được lựa chọn cho phân tích này vì nó cho thấy có nhiều thông tin hơn so với các phần khác của chi Trichoderma [17-19]. Phân tích Bootstrap với 100 lần sao chép Bootstrap đã chứng minh được ba nhánh chính. Trên cơ sở các giá trị bootstrap, 34 Trichoderma spp. có thể được phân thành 5 nhóm. Nhóm 1, phức hợp T. harzianum bao gồm 6 trong các dòng phân lập. Nhóm này được hỗ trợ bởi giá trị bootstrap là 92% có 3 phân nhóm con được hỗ trợ bởi các giá trị bootstrap cao hơn 90%. Nhóm II (Lutea) bao gồm 3 chủng đại diện cho T. brevicompactum. Cụm này được hỗ trợ bởi giá trị bootstrap là 83%. Nhóm IV (Rufa) bao gồm 3 chủng khác nhau bao gồm T. viride, T. ovalisporum và T. erinaceum, nhóm này được hỗ trợ bởi hơn 60% của giá trị bootstrap. Trong trường hợp cụm nhóm III, bao gồm phần Longibrachiatum (H. orientalis) và Pachybasium A (T. asperellum). Trong phân tích này không nhóm nào giữa các chủng được quan sát, và không cho thấy bất kỳ sự hỗ trợ bootstrap nào cho thấy nhánh bất ổn.
Hoạt tính đối kháng và sản sinh Enzyme của các dòng phân lập Trichoderma
Kết quả từ thí nghiệm kép cho thấy tất cả các chủng Trichoderma ức chế sự phát triển của nấm S. rolfsii trên 50%. Hai chủng TGD1 và TWN1 (T. viride và T. harzianum) cho thấy có ý nghĩa thống kê về sự ức chế sự phát triển của tơ nấm (76,3%) đối với S. rolfsii (Bảng 2). Các chủng TND1, TBC1, TCC1, TGN1 và TWC2 (T. erinaceum, T. brevicompactum, T. valisopum, H. lixii và T. harzianum) là những chủng phân lập tốt nhất tiếp theo trong việc ức chế sự phát triển của mầm bệnh, tương ứng là 73,3, 70,8, 72,5, 70,8 và 72,1% so với đối chứng. Dòng phân lập TSD1 (T. inhamatum), TJP1 (H. lixii) và TND1 (T. erinaceum) cho thấy có ý nghĩa thống kê về sự ức chế sự phát triển của nấm C. gloeosporioides trong nuôi cấy kép (Bảng 2). Độ ức chế tăng trưởng của các chủng này lần lượt là 45,6, 43,3 và 42,2% so với nhóm đối chứng. Tương tự trong trường hợp C. capsici, dòng phân lập TWC2 (T. harzianum) và TSD1 (T. inhamatum) cho thấy sự ức chế đáng kể sự phát triển của tơ nấm. Sản xuất chitinase và b-1, 3-glucanase được tóm tắt trong Bảng 3 và 4. Tất cả các chủng cho thấy hoạt tính chitinase và β-1,3-glucanase có ý nghĩa hơn với việc bổ sung các nguồn carbon khác nhau làm chất nền trong môi trường cơ bản. Hoạt tính enzyme chitinase cao nhất được ghi nhận ở T. viride (42.0 U) trong CDB + bột tơ nấm S. rolfsii, sau đó là T. harzianum (40.3 U), T. erinaceum (37.1 U), H. lixii (36.4 U), T. brevicompactum (35.1 U), T. ovalisporum (34.2 U) và Trichoderma sp. (33.2 U). Hoạt tính chitinase trong kiểm tra các dòng phân lập Trichoderma ít liên quan đến việc bổ sung bột tơ nấm C. gloeosporioides và C. capsici vào môi trường cơ bản khi so với S. rolfsii whereas trong khi hoạt tính chitinase cao nhất được ghi nhận ở T. harzianum. he partial substitution of sucrose bằng nguồn cacbon giống nhau trong môi trường cơ bản ảnh hưởng mạnh như hoạt tính β-1,3-glucanase khi so với môi trường cơ bản (CDB). Hoạt tính β-1,3-glucanase cao nhất được ghi nhận ở T. harzianum (22.6, 31.5, 28.6 và 30.2 U) trên CDB, CDB + S. rolsii, CDB + C. gloeosporioides và CDB + C. capsici sau đó là T. viride, T. erinaceum và T. inhamatum. Trong tất cả các nguồn cacbon cải tiến môi trường, bột tơ nấm S. rolsii cảm ứng hoạt tính β-1,3-glucanase và chitinase cao hơn so với C. gloeosporioides và C. capsici.
Ứng dụng của Trichoderma như yếu tố kiểm soát sinh học được hướng đến chủ yếu trong kiểm soát các bệnh ở rễ và trong đất khi có rất ít báo cáo về ứng dụng của nó trong kiểm soát các bệnh ở lá [20]. Điều này xuất phát từ sự hiện diện của Trichoderma trong đất nhưng chỉ xuất hiện tạm thời ở bề mặt lá [21]. Sự đối kháng bởi Trichoderma spp. với nhiều loại đất mang bệnh cây được thực hiện [6, 22, 23]. Sự đối kháng mạnh bởi Trichoderma spp. chống lại vùng đất mang mầm bệnh cây đã được báo cáo [6, 23]. Trichoderma spp. Cũng được biết đến với tiềm năng tạo vách tế bào làm giảm các enzyme bằng cách dùng các vật chất có sẵn trong môi trường tăng trưởng [6]. Các enzyme hydrolytic như β-1,3-glucanase, chitinase, cellulose và proteinase tăng rõ rệt khi Trichoderma spp. tăng trưởng trên môi trường bổ sung tơ nấm đã khử trùng hay các vách tế bào nấm chủ đã được làm sạch [7, 24]. Ulhoa và Peberdy [25] đã tìm thấy các sản phẩm của chitin giảm cũng như quy định sự sinh tổng hợp chitinase ở T. harzianum. Kumar và Gupta [2] báo cáo rằng vách tế bào M. phaseolina và S. rolfsii có glucan và chitin đã cho thấy sự cảm ứng glucanase và chitinase ở tơ nấm trên môi trường cải tiến. Các hoạt tính b-1,3- glucanase và chitinase được phát hiện trong nuôi cấy kép để T. harzianum ký sinh lên R. solani và S. rolfsii và so sánh với mức chất nền hay không có sự hiện diện của mầm bệnh [26]. Trong khảo sát này, hoạt tính enzyme chitinase và β-1,3-glucanase tăng khi thay thế nguồn carbon đặc biệt ở nồng độ 1%. Các phát hiện này phù hợp với Ulhoa và Peberdy [25] khi họ cho rằng hoạt tính chitinase trên bề mặt cơ chất trên 0.5% (w/v) chitin không thể tổng hợp thành chitinase trên môi trường tăng trưởng khi T. harzianum tăng lên nồng độ 1%, trong khi sản phẩm β-1,3-glucanase enzyme tăng đến nồng độ 1% laminarin nhưng giảm ở nồng độ cao hơn. Điều này có thể vì thực tế ở nồng độ đường cao hơn hoạt tính của enzyme này bị ức chế. Mức ức chế của tơ nấm lên mục tiêu gây bệnh nhờ T. erinaceum là ví dụ điển hình như sự kiểm soát sinh học chống lại các tác nhân gây bệnh đã được thử nghiệm.
Lời cảm ơn
Nghiên cứu này được hỗ trợ bởi trợ cấp từ dự án “Ứng dụng của vi sinh vật trong nông nghiệp và các lĩnh vực liên quan” của NBAIM-ICAR, UP, Mau, Ấn Độ. Chúng tôi cảm ơn Trường phòng Giống cây trồng, CARI đã luôn ủng hộ và khuyến khích trong suốt quá trình nghiên cứu.
Bảng 1 Các dòng phân lập Trichoderma được nghiên cứu, định danh và vật chủ
Hình 1 Cây phân loài dựa vào vùng ITS của bộ gen rRNA của 12 dòng phân lập và 22 dòng liên quan ở Trichoderma với số phân lập chỉ ra khoảng cách. Phylogram được tạo ra sau khi dựa vào khoảng cách DNA và phân tích neighbor-joining của dữ liệu. Bootstrap 50% cây đồng thuận đa quy tắc (1000 bản sao) chỉ ra trên các nốt, được tạo ra khi dùng MEGA 3.1
Tài liệu tham khảo:
1. Samuels GJ (1996) Trichoderma: a review of biology and systematics of the genus. Mycol Res 100:923–935
2. Kumar A, Gupta JP (1999) Variation in enzyme activity of tubeconazole tolerant biotypes of Trichoderma viride. Ind Phytopathol 52:263–266
3. Kubicek CP, Penttila ME (1998) Regulation of production ofplant polysaccharide degrading enzymes by Trichoderma. In:Harman GE, Kubicek CP (eds) Trichoderma and Gliocladiumenzymes biological control and commercial applications, vol 2.Taylor and Francis Ltd, London, pp 49–71
4. Sivasithamparam K, Ghisalberti EL (1998) Seconday metabolismin Trichoderma and Gliocladium. In: Kubicek CP, Harman GE(eds) Trichoderma and Gliocladium basic biology taxonomy andgenetics, vol 1. Taylor and Francis Ltd, London, pp 139–191
5. Harman GE (2000) Myth and dogmas of biocontrol; changes inperceptions derived from research on Trichoderma harzianumT-22. Plant Dis 84:377–393
6. Harman GE, Howell CR, Viterbo A, Chet I, Lorito M (2004)Trichoderma species-opportunistic, avirulent plant symbionts.Nat Rev Microbiol 2:43–56
7. Cruz L, Pinter-Toro JA, Benitez T, Llobell A (1995) Purification and characterization of an endo-b-1,3-glucanase from Trichoderma harzianum that is related to its mycoparasitism. J Bacteriol 177:1864–1877
8. Elad Y, Chet I, Henis Y (1982) Degradation of plant pathogenic fungi by Trichoderma harzianum. Can J Microbiol 28:719–725
9. Samuels GJ, Dodd SL, Gams W, Castlebury LA, Petrini O (2002)Trichoderma species associated with the green mold epidemic of commercially grown Agaricus bisporus. Mycologia 94:146–170
10. Raeder U, Broda P (1985) Rapid preparation of DNA from filamentous fungi. Lett Appl Microbiol 1:17–20
11. White TJ, Bruns T, Lee S, Taylor J (1990) Amplification and direct sequencing of fungal ribosomal DNA for phylogenetics. In: Innes MA, Gelfand DH, Sninsky JJ, White TJ (eds) PCR protocols: a guide to methods and applications. Academic, San Diego,CA, pp 315–322
12. Jukes TH, Cantor CR (1969) Evolution of protein molecules. In:Munro HN (ed) Mammalian protein 242 metabolism. Academic,New York, pp 21–132
13. Morton DT, Stroube NH (1955) Antagonistic and stimulatory effect of microorganism upon Sclerotium rolfsii. Phytopathology 45:419–420
14. Reissing JL, Strominger JL, Leloir LF (1955) A modified colorimetric method for estimation of N-acetylamine sugars. J Biol Chem 217:959–966
15. Rifai MA (1969) A revision of the genus Trichoderma. Mycol Pap 116:1–56
16. Bhagat S (2008) Biocontrol potential of Trichoderma spp. from Andaman and Nicobar Islands. PhD thesis, BCKV, West Bengal,India
17. Kuhls K, Lieckfeldt E, Samuels GJ, Kovacs W, Meyer W, Petrini O, Gams W, Borner T, Kubicek CP (1996) Molecular evidence that the asexual industrial fungus Trichoderma reesei is a clonal derivate of the ascomycete Hypocrea jecorina. Proc Natl Acad Sci USA 93:7755–7760
18. Kuhls K, Lieckfeldt E, Samuels GJ, Meyer WC, Kubicek P, Borner T (1997) Revision of Trichoderma sec. Longibrachiatum including related teleomorphs based on analysis of ribosomal DNA internal transcribed spacer regions. Mycologia 89:442–460
19. Ospina-Giraldo MD, Royse DJ, Thon MR, Chen X, Romaine CP (1998) Phylogenetic relationships ofTrichoderma harzianum causing mushroom green mold in Europe and North America to other species of Trichoderma from world-wide sources. Mycologia 90:76–81
20. Blakeman JP, Fokemma NJ (1982) Potential for biological control of plant disease on the phylloplane. Ann Rev Phytopathol 20:167–192
21. Bankole SA (1990) Micro flora associated with cowpea phylloplane and their role in disease development and control. MSc thesis, University of Ibadan, Ibadan
22. Bhagat S, Pan S (2007) Mass multiplication of Trichoderma harzianum on agricultural byproducts and their evaluation against seedling blight of mungbean and collar rot of groundnut. Ind J Agric Sci 77:583–588
23. Bhagat S, Pan S (2008) Variability in production of extra cellular hydrolytic enzymes by Trichoderma spp. and induction of disease resistance in gram (Ciccer arietinum). J Biol Conserv 22:57–66
24. Carasolio CA, Gutierrez A, Jimenez B, Van Monkngu M, Herrera Estrella A (1994) Primary structures and expression pattern of the 33 kDa chitinase gene from the mycoparasitic fungus Trichoderma harzianum. Proc Natl Acad Sci USA 91:10903–10907
25. Ulhoa CJ, Peberdy JF (1991) Regulation of chitinase synthesis in Trichoderma harzianum. J Gen Microbiol 137:2163–2169
26. Elad Y, Chet I (1983) Improved selective media for isolation of Trichoderma spp. and Fusarium spp. Phytoparasitica 11:55–58
Trâm Anh
SBC Scientific